(新SOTA)UNETR++:轻量级的、高效、准确的共享权重的3D医学图像分割
0 Abstract
由于Transformer模型的成功,最近的工作研究了它们在3D医学分割任务中的适用性。在Transformer模型中,与基于局部卷积的设计相比,自注意力机制是努力捕获远程依赖性的主要构建块之一。然而,self-attention操作具有平方复杂性,这被证明是一个计算瓶颈,特别是在三维医学成像中,输入是具有大量切片的3D图像。在本文中,我们提出了一种名为UNETR++的3D医学图像分割方法,该方法既提供高质量的分割masks,又在参数和计算成本方面具有效率。我们设计的核心是引入一种新颖的高效配对注意(EPA)块,该块使用一对基于空间和通道注意的相互依赖的分支来有效地学习空间和通道方面的判别特征。我们的空间注意力公式是高效的,相对于输入的序列长度具有线性复杂度。为了实现以空间和通道为中心的分支之间的通信,我们共享query和key映射函数的权重,这些函数提供了互补的好处(配对注意力),同时还减少了整体网络参数。我们对 Synapse、BTCV 和 ACDC、BRaTs和Decathlon-Lung五个基准进行了广泛的评估,揭示了所提出的贡献在效率和准确性方面的有效性。在Synapse数据集上,我们的UNETR++以87.2% 的Dice相似度得分创下了新的SOTA水平,同时与文献中最好的现有方法相比,在参数和FLOPs方面都明显高效,减少了71%以上。
1 Introduction
3D分割是医学成像中的一个基本问题,具有众多应用,包括用于诊断目的的肿瘤识别和器官定位。该任务通常通过使用类似编码器-解码器架构的U-Net来解决,其中编码器生成3D图像的分层低维表示,解码器将此学习表示映射到体素分割。早期的基于CNN的方法分别在编码器和解码器中使用卷积和反卷积,但可能由于其有限的局部感受野而难以获得准确的分割结果。另一方面,基于Transformer的方法本质上是全局的,并且最近以增加模型复杂性为代价展示了具有竞争力的性能。
最近,几项工作 [Swin UNETR、UNETR、nnformer] 探索了设计混合架构以结合局部卷积和全局注意力的优点。虽然一些方法[UNETR] 使用基于Transformer的编码器和卷积解码器,但其他方法[Swin UNETR、nnformer] 旨在为编码器和解码器子网络设计混合块。然而,这些工作主要集中在提高分割精度,这反过来又在参数和FLOPs方面大大增加了模型的大小,导致鲁棒性不尽如人意。我们认为,这种不令人满意的鲁棒性可能是由于它们低效的self-attention设计,这在3D医学图像分割任务中变得更加成问题。此外,这些现有方法没有捕捉到可以提高分割质量的空间和通道特征之间的显式依赖关系。在这项工作中,我们的目标是在一个统一的框架中同时提高分割精度和模型效率。
贡献: 我们提出了一种用于3D医学图像分割的高效混合层次结构,名为 UNETR++,它力求在参数和FLOP方面实现更好的分割精度和效率。基于最近的UNETR框架,我们提出的UNETR++分层方法引入了一种新颖的高效配对注意力 (EPA) 块,通过在两个分支中同时使用空间和通道注意力有效的捕获丰富的相互依赖的空间和通道特征。我们在EPA中的空间注意力将key和value投射到固定的低维空间,使自注意力计算与输入tokens的数量成线性关系。另一方面,我们的通道注意力通过在通道维度中执行query和key之间的点积运算来强调通道特征图之间的依赖关系。此外,为了捕获空间和通道特征之间的强相关性,query和key的权重在分支之间共享,这也有助于控制网络参数的数量。相反,value的权重保持独立,以强制在两个分支中学习互补特征。
我们通过对三个基准进行综合实验来验证我们的UNETR++方法:Synapse、BTCV 和 ACDC。定性和定量结果都证明了UNETR++的有效性,与文献中的现有方法相比,在分割精度和模型效率方面具有更好的性能。在Synapse上,与基线 UNETR 相比,我们的 UNETR++ 实现了高质量的分割Mask(见图 1 左),在Dice相似度得分方面的绝对增益为8.9%,同时显着降低了模型复杂性,参数减少了54%,FLOPs减少了 37%。此外,UNETR++优于现有最好的 nnFormer方法,在参数和 FLOP 方面都有相当大的减少(参见右图 1)。
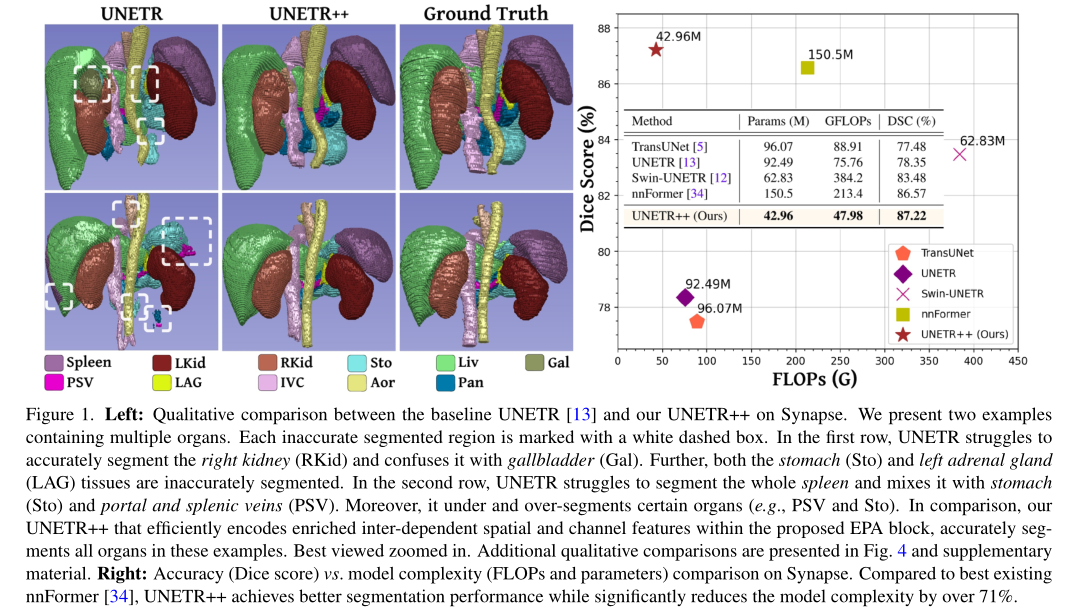
图1. 左图: 基线 UNETR [13] 和我们的 UNETR++ 在Synapse 之间的定性比较。我们展示了两个包含多个器官的例子。每个不准确的分割区域都用白色虚线框标记。在第一行中,UNETR 难以准确分割右肾 (RKid) 并将其与胆囊 (Gal) 混淆。此外,胃 (Sto) 和左肾上腺 (LAG) 组织的分割不准确。在第二行,UNETR 努力分割整个脾脏并将其与胃 (Sto) 以及门静脉和脾静脉 (PSV) 混合。此外,它对某些器官(例如,PSV 和 Sto)进行了过度分割。相比之下,我们的UNETR++在提出的 EPA 块中有效地编码了丰富的相互依赖的空间和通道特征,准确地分割了这些示例中的所有器官。图像最好放大看。其他定性比较显示在图4和补充材料中。右图: Synapse 上的准确性(Dice 分数)与模型复杂性(FLOP 和参数)比较。与现有的最佳 nnFormer相比,UNETR++ 实现了更好的分割性能,同时将模型复杂度显着降低了 71% 以上。
2 Related Work
基于 CNN 的分割方法: 自从引入U-Net设计以来,几种基于 CNN 的方法[D-Unet、Unet3+、Unet++等] 已经针对各种医学图像分割任务扩展了标准 U-Net 架构。
在 3D 医学图像分割 [3D UNet,Automatic multi-organ segmentation on abdominal ct with dense v-networks(TMI),V-net, MedT] 的情况下,整个体积图像通常作为 2D切片序列进行处理。几项工作探索了层次框架来捕获上下文信息。 VNet提出通过将体积下采样到较低分辨率来使用体积图像的3D表示,以保留有益的图像特征。 3D Unet通过将2D操作替换为对应的3D操作,从稀疏注释的体积图像中学习,将 U-Net 架构扩展到体积分割。 Isensee 等人介绍了一个名为 nnUNet 的通用分割框架,它可以自动配置架构以在多个尺度上提取特征。
Hierarchical 3d fully convolutional networks for multi-organ segmentation.提出了一个多尺度 3D 全卷积网络来学习来自多器官分割的不同分辨率的表示。此外,文献中已经做出多项努力,使用例如图像金字塔 、大卷积核、扩张卷积和可变形卷积在基于 CNN 的框架内对整体上下文信息进行编码。
基于 Transformers 的分割方法: Vision Transformers (ViTs) 最近受到欢迎,因为它们能够对远程依赖项进行编码从而在各种视觉任务上取得有希望的结果,包括分类和检测。 Transformer 架构中的主要构建块之一是自注意力操作,它对图像块序列之间的交互进行建模,从而学习全局关系。最近很少有工作探索如何减轻 transformer框架内标准自注意力操作的复杂性问题。然而,这些最近的工作大多主要集中在分类问题上,并没有针对密集预测任务进行研究。
在医学图像分割的背景下,最近的工作很少研究纯Transformer设计。Convolution-free medical image segmentation using transformers[MICCAI 2021]提出将体积图像划分为 3D 块,然后将其展平以构建1D嵌入并传递给主干以进行全局表示。 Swin Unet介绍了一种用于2D医学图像分割的具有移位窗口的架构。在这里,图像被分成小块并送入U形编码器解码器以进行局部-全局表示学习。
混合分割方法: 除了纯 CNN 或基于变换器的设计之外,最近的几项工作探索了混合架构以结合卷积和自注意力操作以实现更好的分割。 TransFuse提出了一种带有BiFusion模块的并行 CNN-transformer 架构,以融合编码器中的多级特征。 MedT在 self-attention 中引入了门控位置敏感轴向注意机制来控制编码器中的位置嵌入信息,而解码器中的 ConvNet 模块产生分割模型。 TransUNet结合了transformer和 U-Net 架构,其中变换器对来自卷积特征的嵌入图像块进行编码,解码器将上采样编码特征与高分辨率 CNN 特征相结合以进行定位。
Ds-transunet利用基于 Swin transformer的双尺度编码器来处理多尺度输入,并通过自注意力对来自不同语义尺度的局部和全局特征表示进行编码。 Hatamizadeh 等人介绍了一种 3D 混合模型 UNETR,它将transformer的远程空间依赖性和 CNN 的归纳偏置结合到一个“U 形”编码器解码器架构中。 UNETR 中的变换器块主要用于编码器中以提取固定的全局表示,然后在多个分辨率下与基于 CNN 的解码器合并。 Zhou 等人介绍了一种名为 nnFormer 的方法,它采用了 Swin-UNet架构。在这里,卷积层将输入扫描转换为 3D 补丁,并引入基于体积的自注意力模块来构建分层特征金字塔。
在实现有前途的性能的同时,与 UNETR 和其他混合方法相比,nnFormer 的计算复杂度要高得多。
我们的方法: 如上所述,与纯 CNN 和基于transformer的对应方法相比,最近的混合方法(例如 UNETR和 nnFormer)实现了改进的分割性能。
然而,我们注意到,这种通过这些混合方法提高分割精度是以更大的模型为代价的(在参数和 FLOP 方面),这可能进一步导致不令人满意的鲁棒性。例如,与现有最好的基于 CNN 的 nnUNet 相比,UNETR 实现了良好的精度,但包含的参数多了2.5倍。此外,nnFormer 获得了优于 UNETR 的性能,但参数进一步增加了 1.6 倍,FLOPs 增加了 2.8 倍。此外,我们认为,上述这些混合方法难以有效地捕获特征通道之间的相互依赖关系,以获得对空间信息和通道间特征依赖关系进行编码的丰富特征表示。在这项工作中,我们着手在一个统一的混合分割框架中共同解决上述问题。
3 Method
动机: 为了实现我们的方法,我们首先区分了在设计高效而准确的混合框架时要考虑的两个理想属性。
Efficient Global Attention: 如前所述,大多数现有的混合方法采用自注意力操作,在tokens数量方面具有平方复杂性。在3D医学分割的情况下,这在计算上是昂贵的,并且在混合设计中交错窗口注意力和卷积组件时变得更成问题。与这些方法不同,我们认为跨特征通道而不是体积维度计算自注意力有望将体积维度的复杂性从平方降低到线性。此外,通过将key和value的空间矩阵投影到低维空间中,可以有效地学习空间注意力信息。
Enriched Spatial-channel Feature Representation: 大多数现有的混合3D医学图像分割方法通常通过注意力计算来捕获空间特征,并以编码不同通道特征图之间的相互依赖关系的形式忽略通道信息。有效地结合空间维度中的相互作用和通道特征之间的相互依赖性有望提供丰富的上下文空间通道特征表示,从而改进mask预测。
3.1.总体架构
图 2 展示了我们的 UNETR++ 架构,包括分层编码器-解码器结构。
我们的 UNETR++ 框架基于最近推出的 UNETR,在编码器和解码器之间具有跳过连接,然后是卷积块(ConvBlocks)来生成预测掩码。我们的 UNETR++ 没有在整个编码器中使用固定的特征分辨率,而是采用分层设计,其中特征分辨率在每个阶段逐渐降低两倍。在我们的 UNETR++ 框架中,编码器有四个阶段,其中第一阶段包括Patch嵌入以将体积块输入划分为3D补丁,然后是我们新颖的高效配对注意 (EPA) 块。
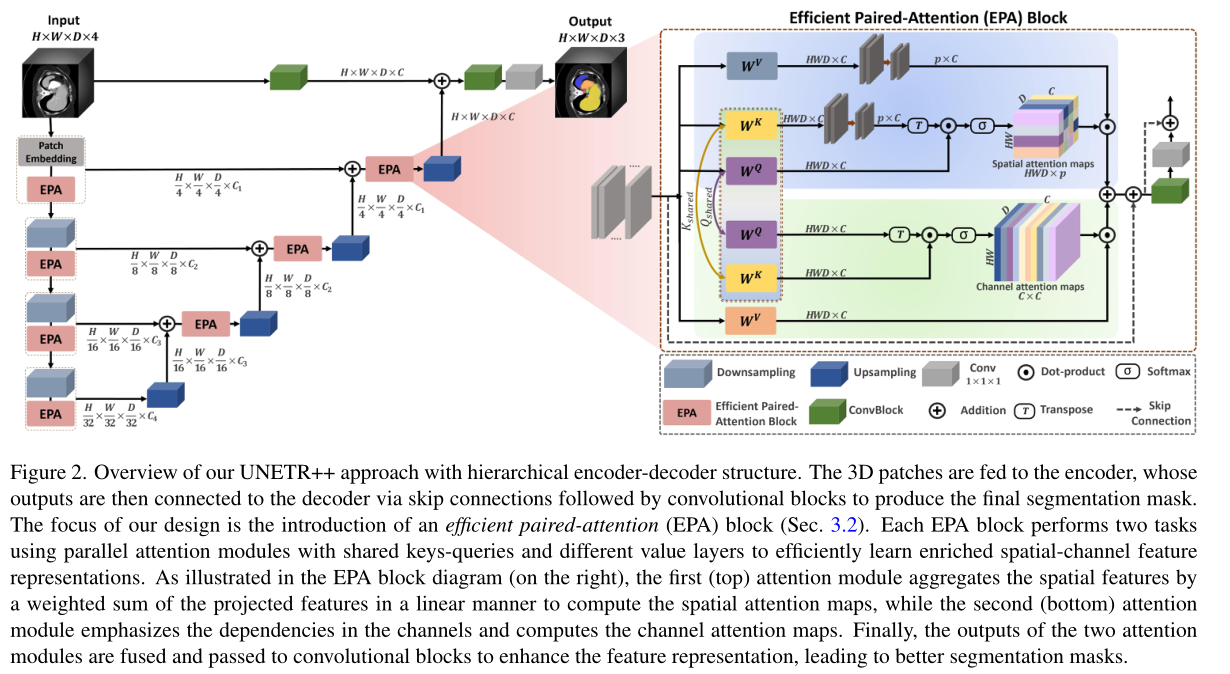
图 2. 我们的具有分层编码器-解码器结构的 UNETR++ 方法概述。3D的Patch被馈送到编码器,然后编码器的输出通过跳过连接连接到解码器,然后是卷积块以产生最终的分割掩码。我们设计的重点是引入高效的配对注意 (EPA) 块(第 3.2 节)。每个 EPA 块使用具有共享keys-queries和不同值层的并行注意模块执行两项任务,以有效地学习丰富的空间通道特征表示。如EPA框图(右侧)所示,第一个(顶部)注意力模块通过线性方式对投影特征进行加权和来聚合空间特征,以计算空间注意力图,而第二个(底部)注意力模块模块强调通道中的依赖关系并计算通道注意力图。最后,两个注意模块的输出被融合并传递给卷积块以增强特征表示,从而产生更好的分割Mask。
在块嵌入中,我们将每个3D输入(体积)x ∈ RH×W ×D 分成不重叠的块 xu ∈ RN×(P1,P2,P3) ,其中 (P1, P2, P3) 是每个块的分辨率patch 和 N = ( H/P1 × W/P2 × D/P3 ) 表示序列的长度。然后,将patch投影到C通道维度,生成大小为 H/P1 × W/P2 × D/P3 ×C 的特征图。我们使用与nnformer中相同的Patch分辨率 (4, 4, 2)。对于每个剩余的编码器阶段,我们采用使用非重叠卷积的下采样层将分辨率降低两倍,然后是 EPA 块。
在我们提出的 UNETR++ 框架中,每个 EPA 块包含两个注意模块,通过使用共享keys-queries方案对空间和通道维度的信息进行编码来有效地学习丰富的空间通道特征表示。编码器阶段通过跳跃连接与解码器阶段连接,以合并不同分辨率的输出。这使得能够恢复在下采样操作期间丢失的空间信息,从而预测更精确的输出。与编码器类似,解码器也包括四个阶段,其中每个解码器阶段都包含一个上采样层,使用反卷积将特征图的分辨率提高两倍,然后是 EPA 块(最后一个解码器除外)。每两个解码器级之间的通道数量减少两倍。因此,最后一个解码器的输出与卷积特征图融合以恢复空间信息并增强特征表示。
然后将生成的输出馈入 3×3×3 和 1×1×1 卷积块以生成体素方面的最终mask预测。接下来,我们详细介绍我们的 EPA 块。
3.2. Efficient Paired-Attention Block
提出的EPA块执行高效的全局注意力并有效地捕获丰富的空间通道特征表示。 EPA 块包括空间注意力和通道注意力模块。空间注意模块将自注意力的复杂性从平方降低到线性。另一方面,通道注意力模块有效地学习了通道特征图之间的相互依赖关系。EPA块基于两个注意模块之间的共享keys-queries方案,相互通知以生成更好、更有效的特征表示。这可能是由于通过共享key和query但使用不同的value层来学习互补特征。
如图 2(右)所示,由大小为 H/4 × W/4 × D/2 × C 的块嵌入 x 生成的特征图被直接馈送到连续的 EPA块中,然后是三个编码器阶段。 Q和K线性层的权重在两个注意力模块之间共享,并且每个注意力模块使用不同的 V 层。两个注意模块计算如下:
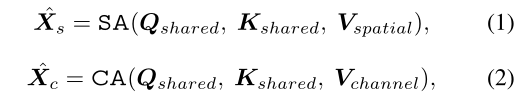
Xs、Xc分别表示空间和通道注意力图,SA是空间注意力模块,CA是通道注意力模块。 Qshared、Kshared、Vspatial、Vchannel分别是共享query、共享key、空间value层、通道value层的矩阵。
空间注意: 我们在这个模块中努力通过将复杂度从 O(n2) 降低到 O(np)来有效地学习空间信息,其中n是token的数量,p是投影向量的维度,其中 p<<n。给定形状为 HW D×C 的归一化张量 X,我们使用三个线性层计算Qshared、Kshared和Vspatial投影,得到 Qshared=WQX、Kshared=WKX 和 Vspatial=WV X,维度为 HW D×C,其中W Q、W K 和 W V 分别是 Qshared、Kshared 和 Vspatial 的投影权重。然后,我们执行三个步骤。首先,将 Kshared和 Vspatial层从 HW D × C 投影到形状为 p × C 的低维矩阵。其次,通过将 Qshared层乘以投影的Kshared的转置来计算空间注意力图,然后用 softmax 来测量每个特征与其余空间特征之间的相似性。第三,将这些相似性乘以投影的V空间层,以生成形状为HW D×C的最终空间注意力图。空间注意力定义如下:

Channel Attention: 该模块通过在通道value层和通道注意力图之间的通道维度中应用点积运算来捕获特征通道之间的相互依赖关系。使用空间注意力模块的相同 Qshared 和 Kshared,我们计算通道的value层以使用线性层学习互补特征,产生 Vchannel = WVX,维度为 HWD×C,其中 WV 是 Vchannel 的投影权重。通道注意力定义如下:
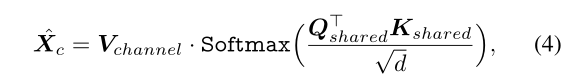
其中,Vchannel、Qshared、Kshared分别表示通道value层、共享query和共享key,d是每个向量的大小。
最后,我们执行总和融合并通过卷积块转换两个注意力模块的输出以获得丰富的特征表示。 EPA 块的最终输出X得到为:

3.3.损失函数
我们的损失函数基于常用的soft Dice损失和交叉熵损失的总和,以同时利用这两种互补损失函数的好处。它被定义为:
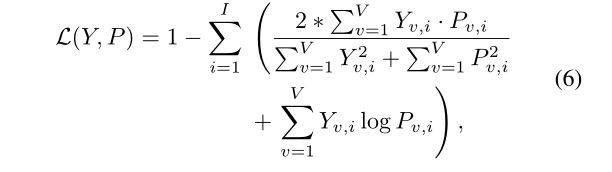
其中,I 表示类数; V 表示体素数; Yv,i 和 Pv,i 分别表示类 i 在体素 v 处的groud truth和输出概率。
4.实验
4.1 实验设置
我们在五个数据集上进行了实验:Synapse for Multi-organ CT Segmentation, BTCV for Multi-organ CT Segmentation, ACDC for Automated Cardiac Diagnosis, Brain Tumor Segmentation (BraTS) Medical Segmentation decathon - lung。
数据集:
用于多器官 CT 分割的Synapse 数据集来自 MICCAI Multi-Atlas Labeling Beyond the Cranial Vault challenge(MALBCV,多图谱标记超越颅穹窿挑战),由 30 名受试者的 8 个器官的腹部 CT 扫描组成。与以前的方法一致,我们遵循Transunet中使用的拆分并在 18 个样本上训练我们的模型并在其余 12 个案例上进行评估。我们使用Dice相似系数 (DSC) 和 95% Hausdorff 距离 (HD95) 报告模型在 8 个腹部器官上的表现:脾脏、右肾、左肾、胆囊、肝脏、胃、主动脉和胰腺。
用于多器官 CT 分割的 BTCV数据集包含30个用于训练/验证的对象和 20 个用于腹部 CT 扫描测试的对象。它由 13 个器官组成,包括 Synapse 数据集的所有 8 个器官,以及食道、下腔静脉、门静脉和脾静脉、右肾上腺和左肾上腺。该数据集是在范德比尔特大学医学中心的临床放射科医生的监督下手动注释的。每次扫描包含 80 到 225 个切片,其中每个切片的空间大小为 512×512 像素,厚度为1到 6mm。与nnformer一致,我们对每个独立扫描CT图片通过将 [-1000, 1000]HU范围内的强度归一化到[0, 1],以128×128×64的Patch裁剪和 [0.76, 0.76, 3] 的间距进行预处理。 我们报告了所有 13 个腹部器官的 Dice 相似系数 (DSC)。
ACDC for Automated Cardiac Diagnosis数据集包含从真实临床检查中收集的100名患者的心脏MRI图像,以及右心室(RV)、左心室(LV) 和心肌 (MYO) 的相应分割注释。从正常患者、患有心肌梗塞、扩张型心肌病、肥厚型心肌病和异常右心室的患者中收集样本。与nnformer一致,我们将数据分成 70 个训练图像、10 个验证样本,并对剩余的 20 个样本进行评估。我们报告了三个类别的 DSC。 与nnformer一致我们将数据分为70、10和20个训练、验证和测试样本。我们报告了这三个类的DSC。
BraTS由484张MRI图像组成,每张图像由FLAIR、T1w、T1gd和T2w四个通道组成。我们将数据集分成80:5:15的比例进行训练、验证和测试,并在测试集上报告。目标分类为全肿瘤 (WT)、增强肿瘤(ET)和肿瘤核心(TC)。
肺数据集包含63个CT数据,用于一个两类问题,目标是从背景中分割肺癌。我们将数据分成80:20的比例进行训练和验证。
评估指标: 我们根据两个指标衡量模型的性能:Dice相似度分数 (DSC) 和 95% Hausdorff 距离 (HD95)。 DSC测量体积分割预测与Ground Truth体素之间的重叠,其定义如下:
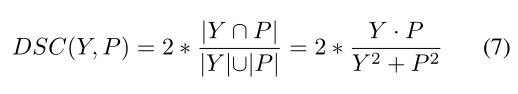
其中,Y 和 P 分别表示所有体素的Ground Truth和输出概率。
HD95通常用作基于边界的度量来测量体积分割预测的边界与Ground Truth的体素之间距离的第 95 个百分位数。它的定义如下:
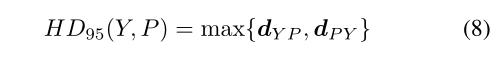
其中,dY P 是预测体素与真实体素之间的最大 95% 距离,dP Y 是真实体素与预测体素之间的最大 95% 距离。
实施细节: 我们在 Pytorch v1.10.1 中实施我们的方法并使用 MONAI 库。为了与基线 UNETR 和 nnFormer 进行公平比较,我们使用相同的输入大小、预处理策略并且没有额外的训练数据。这些模型使用单个A100 40GB GPU进行训练,输入 3D 块大小为128 × 128 × 64,用于1k个epochs,学习率为0.01,权重衰减为 3e−5。
此外,我们报告了BTCV的输入大小为96 × 96 × 96和patch分辨率为 (4, 4, 4) 的结果,其中模型训练了 5k个epochs,学习率为 1e-4。具体来说,输入数据在训练过程中被分成不重叠的块,用于通过反向传播学习分割图。在训练期间,我们对 UNETR、nnFormer 和我们的 UNETR++ 应用相同的数据增强。
此外,与 nnFormer 方法一样,我们也使用相同的深度监督方案并在训练期间计算多种分辨率的损失。我们在推理过程中使用重叠率为 0.5 的滑动窗口。更多细节在补充材料中。代码和模型将公开发布。
4.2.基线比较
表2显示了将提出的贡献整合到基线 UNETR [13] 中对 Synapse 的影响。在除了Dice相似系数 (DSC) 之外,我们还根据参数和 FLOP 报告了模型的复杂性。在所有情况下,我们都会根据单个模型的准确性来报告性能。如前所述,UNETR++ 是一种分层架构,在每个阶段后将编码器的特征图下采样两倍。因此,该模型包括四个编码器阶段和四个解码器阶段。我们的 UNETR++ 的这种分层设计通过将参数从 92.49M 减少到 16.60M 并将 FLOP 从 75.76G 减少到 30.75G,同时与基线相比保持78.29% 的 DSC,仍然具有可比性,从而显着降低模型复杂性。在我们的 UNETR++ 编码器中引入 EPA 块可显著提高性能,DSC 绝对增益比基线高 6.82%。通过在解码器中集成 EPA 块进一步提高了性能。与基线相比,我们最终的 UNETR++ 在编码器和解码器中采用新型 EPA 块进行分层设计,使 DSC 显着提高 8.87%,同时将模型复杂性在参数和 FLOPs 中显着降低 54% 和 37%。我们进一步进行了一项实验,以评估我们提出的EPA块内的空间和通道注意力。采用空间和通道注意力显着提高性能,比基线提高DSC,分别为86.42% 和86.39%。在我们的 EPA 块中结合空间和通道注意力导致 DSC 进一步提高 87.22%。
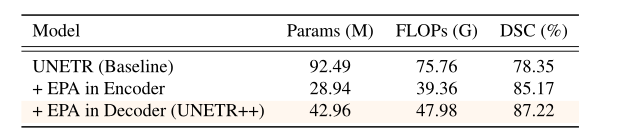
表 2. Synapse 的基线比较。我们根据分割性能 (DSC) 和模型复杂性(参数和 FLOP)显示结果。为了公平比较,所有结果都是使用相同的输入大小和预处理获得的。在我们的分层设计的编码器中集成 EPA 块将分割性能提高到 85.17%。通过在解码器中引入 EPA 块,结果进一步提高到 87.22%。我们的 UNETR++ 在编码器和解码器中均采用新颖的 EPA 块,在 DSC 中实现了 8.87% 的绝对增益,同时还显着降低了模型复杂性。
图 3 显示了基线和我们的 UNETR++ 在 Synapse 数据集上的定性比较。我们放大了几个病例的不同器官(第一行用绿色虚线框标记)。在第一列中,基线难以分割下腔静脉和主动脉。在第二列中,当它们彼此相邻时,它会混淆相同的两个器官。在最后两列中,基线欠切左肾、脾脏和胃,而过切胆囊。相比之下,UNETR++ 通过准确分割所有器官来提高性能。
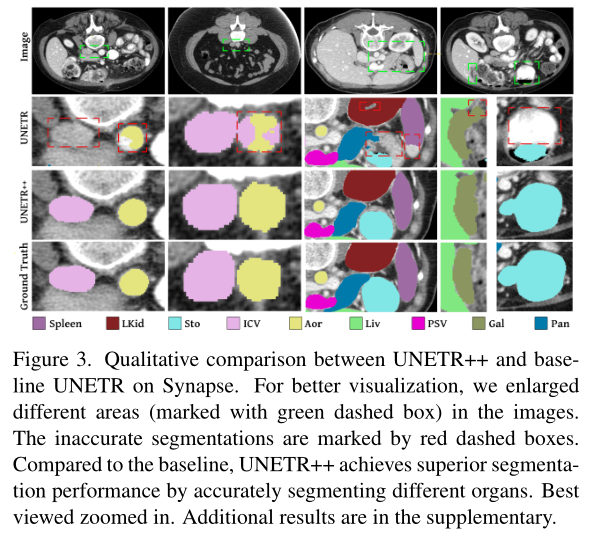
图 3. UNETR++ 和 Synapse 上的基线 UNETR 之间的定性比较。为了更好的可视化,我们放大了图像中的不同区域(用绿色虚线框标记)。不准确的分割用红色虚线框标记。与baseline相比,UNETR++通过准确分割不同器官实现了优越的分割性能。最佳观看放大。附加结果在补充中。
4.3.SOTA比较
**Synapse数据集:**表1 显示了多器官Synapse数据集上的结果。我们使用 DSC 和 HD95 指标报告腹部器官的分割性能。此外,我们报告了每种方法的参数和 FLOP 方面的模型复杂性。分割性能以单一模型精度报告,并且不使用任何预训练、模型集成或额外的数据,纯基于 CNN 的 U-Net方法实现了 76.85% 的 DSC。在现有的基于混合 transformers-CNN 的方法中,UNETR和 Swin-UNETR分别实现了 78.35% 和 83.48% 的 DSC。在这个数据集上,nnFormer获得了优于其他现有作品的性能。我们的 UNETR++ 通过实现 87.22% 的 DSC 优于 nnFormer。此外,就 HD95 指标而言,UNETR++ 比 nnFormer 获得了 3.1% 的绝对误差减少。值得注意的是,UNETR++ 通过在参数和 FLOPs 方面将模型复杂性显着降低 71% 以上,从而实现了分割性能的提高。
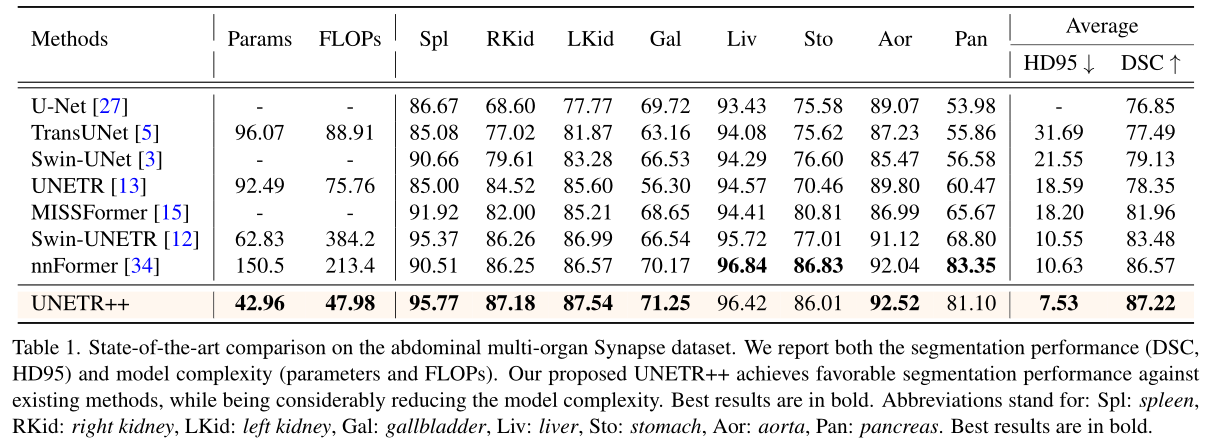
表 1. 腹部多器官Synapse多器官分割数据集SOTA比较。我们报告了分割性能(DSC、HD95)和模型复杂性(参数和FLOP)。我们提出的 UNETR++ 实现了对现有方法有利的分割性能,同时大大降低了模型的复杂性。最佳结果以粗体显示。缩写代表:Spl:脾脏,RKid:右肾,LKid:左肾,Gal:胆囊,Liv:肝,Sto:胃,Aor:主动脉,Pan:胰腺。最佳结果以粗体显示。
图 4 显示了 UNETR++与现有方法在腹部多器官分割的定性比较。在这里,不准确的分割用红色虚线框标记。在第一行中,我们观察到现有方法难以通过在 UNETR 和 Swin UNETR 的情况下对胃进行欠分割或在 nnFormer 的情况下将其与脾混淆来准确分割胃。相比之下,我们的 UNETR++ 准确地分割了胃。此外,现有方法无法完全分割第二行中的右肾。相比之下,我们的 UNETR++ 准确地分割了整个右肾,这可能是由于通过丰富的空间通道表示来学习上下文信息。此外,UNETR++ 平滑地划定了脾脏、胃和肝脏之间的界限。在第三行,UNETR 混淆了胃和胰腺。另一方面,Swin UNETR 和 nnFormer 分别切下胃和左肾上腺。在这些示例中,UNETR++ 准确地分割了所有器官,并更好地描绘了边界。
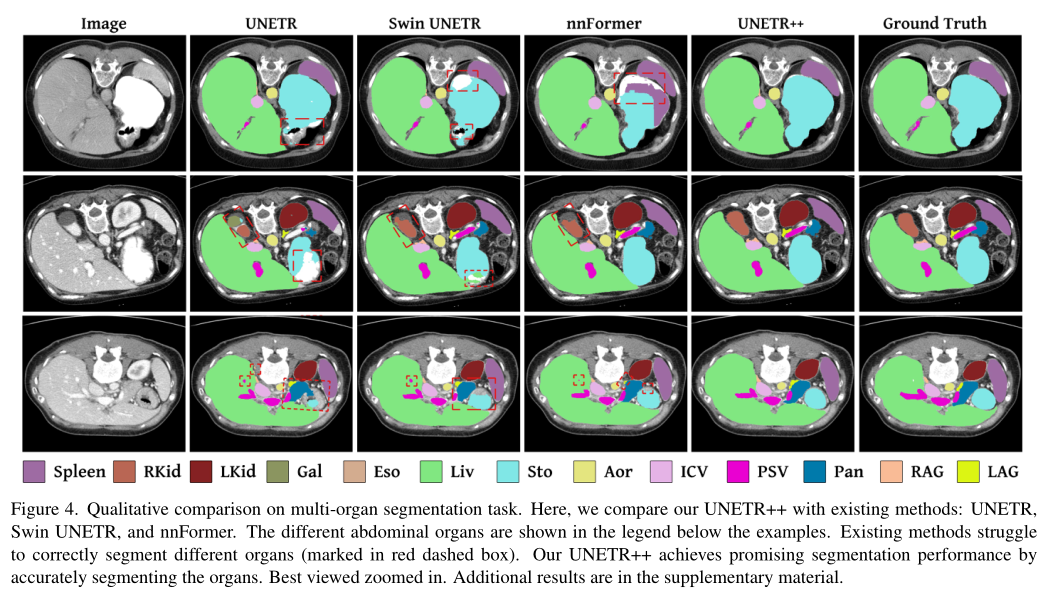
图 4. 多器官分割任务的定性比较。在这里,我们将我们的 UNETR++ 与现有方法进行比较:UNETR、Swin UNETR 和 nnFormer。示例下方的图例中显示了不同的腹部器官。现有方法很难正确分割不同的器官(用红色虚线框标记)。我们的 UNETR++ 通过准确分割器官实现了有前途的分割性能。最佳视图放大。补充材料中有其他结果。
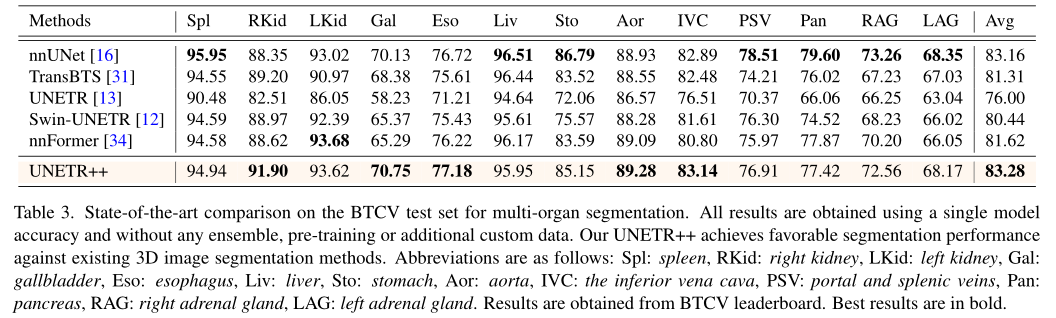
**BTCV 数据集:**表3 展示了在 BTCV 测试集上的比较。在这里,所有结果都基于单个模型的准确性,没有任何集成、预训练或额外数据。我们报告所有 13 个器官的结果以及所有器官的相应平均性能。在现有工作中,UNETR 和 SwinUNETR 实现了 76.0% 和 80.44% 的平均 DSC。在现有方法中,nnUNet 获得了 83.16% 的平均 DSC 性能,但需要 358G FLOPs。相比之下,UNETR++ 通过实现 83.28% 的平均 DSC 而优于 nnUNet,同时需要显着更少的 31G FLOP。
**ACDC 数据集:**选项卡。图 4 显示了 ACDC 的比较。在这里,所有结果均以单一模型精度报告,并且不使用任何预训练、模型集成或其他数据。UNETR和nnFormer的DSC均值分别为86.61%和92.06%。UNETR++取得了较好的性能,平均DSC为92.83%。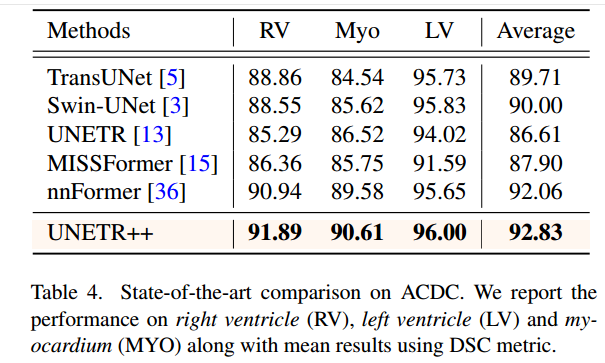
BRaTs数据集: 表5显示了分割性能、模型复杂性和推理时间。为了公平的比较,我们使用相同的输入大小和预处理策略。我们比较了Quadro RTX 6000 24 GB GPU和32核 Intel®Xeon® 4215 CPU的速度。这里,推理时间是使用1×128×128×128输入大小的BRaTs的平均正向通过时间。与最近基于Transformer的方法相比,我们的UNETR++实现了良好的性能,同时以更快的推理速度运行,并且需要更少的GPU内存。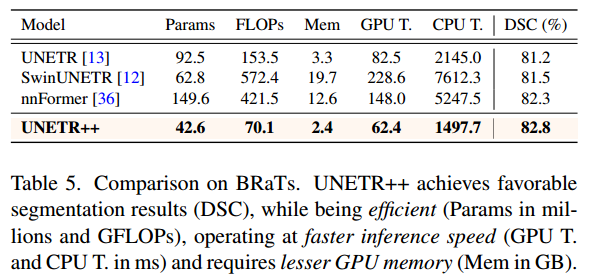
**肺部数据集:**我们评估UNETR++和其他SOTA模型对肺癌分割任务。UNETR++的平均DSC达到80.68%,比现有方法的分割性能更好。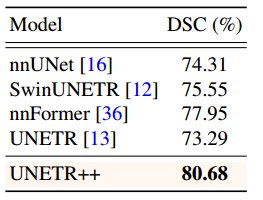
5 结论
我们提出了一种名为 UNETR++ 的分层方法,用于 3D 医学分割。我们UNETR++ 引入了一个有效的成对注意力 (EPA) 块,通过使用空间和通道注意力来编码丰富的相互依赖的空间和通道特征。在 EPA 块中,我们共享query和Key映射函数的权重,以更好地在空间和通道分支之间进行通信,提供互补优势并减少参数。
与现有方法相比,我们的 UNETR++ 在五个数据集上取得了良好的分割结果,同时显着降低了模型的复杂性。
最后强调一下,EPA Block,它的K Q的权重是共享的,为什么叫做Pair呢?
因为它有两个分支,一个是对空间(要算出每个pixel像素点的attention map),另一个是对通道(每一个channel 的attention map),所以说上面是HWD×p的attention map,下面是C×C
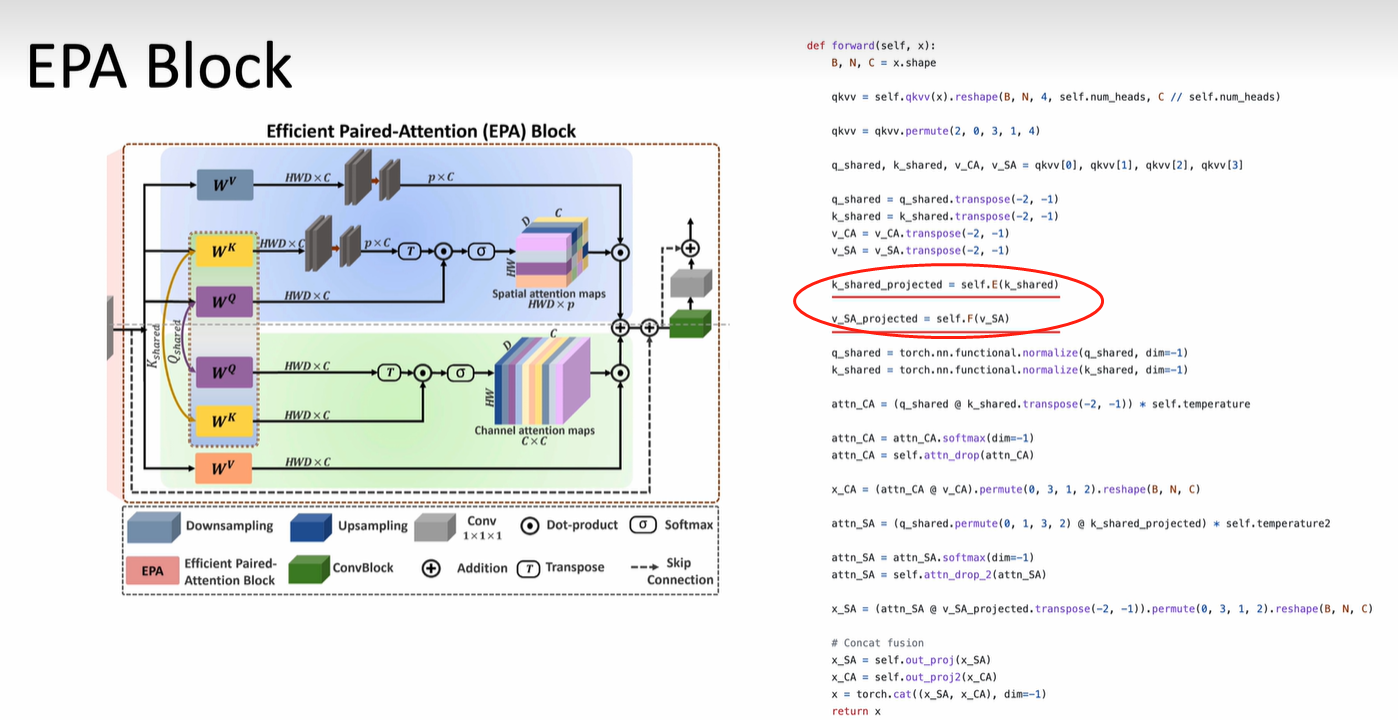
上面为什么要×p呢,按理来说是HWD×HWD,结合代码看,这里做了一个project,把K的HWD的维度映射到p,还有v的维度也映射到p,所以QK计算完就是HWD×p,和p×C的V计算完又变为HWD×C;同理下半部分,最后也能得到HWD×C的feature,两个分支会concat起来,最后得到一个特征。
所以其中做的一个线性映射和share会极大的减少参数量,同时还对Spatial和Channel wise做两种不同的attention也能提高一定的准确率。